理解复杂数据集之间的差异和相似之处是处理数据时经常遇到的一个有趣挑战。将这个问题形式化的一种方法是将每个数据集视为一个图,即项目如何相互关联的数学模型。图被广泛用于模拟对象之间的关系——互联网图连接相互引用的页面,社交图将朋友联系在一起,分子图连接相互结合的原子。
图形是离散的对象,可以模拟许多不同类型的数据之间的关系,包括网页(左)、社交连接(中)或分子(右)。
一旦有了多个图的集合,通常就会希望将每个图的一些属性作为一个集合来预测(即每个图一个标签)。例如,考虑从结构预测蛋白质功能的任务:这里的每个数据集都是一种蛋白质,预测任务是最终结构是否编码酶。由于人们希望模型能够真正计算预测,因此我们需要一种能够让我们概括不同蛋白质结构的表示。理想情况下,人们希望有一种将图表示为向量的方法,而无需昂贵的标签。随着图的大小增加,问题变得越来越困难——在分子情况下,人类拥有一些关于其属性的知识,然而,推理更大、更复杂的数据集变得越来越困难。
在本文中,我们重点介绍了图表示学习领域的一些最新进展,即“近似时只需使用 SLaQ:网络规模图的精确谱距离”(发表于WWW'20),该论文改进了我们早期研究“ DDGK:学习深度发散图核的图表示”(发表于WWW'19)的可扩展性。SLaQ 引入了一种扩展计算以近似某一类图统计数据的方法,可以快速有效地表征大型图。我们也很高兴地宣布,我们已经在 Google Research GitHub 存储库中发布了这两篇论文的图嵌入代码。
图相似性的完全无监督学习
在我们 2019 年的论文中,我们展示了无需领域知识或监督就可以学习图相似性的表示。我们提出了深度发散图核(DDGK),这是一种无监督方法,用于学习图上的表示,这些表示对它们之间的相似性映射进行编码。与以前的工作不同,我们的无监督方法联合学习节点表示、图表示和基于注意的图间对齐。
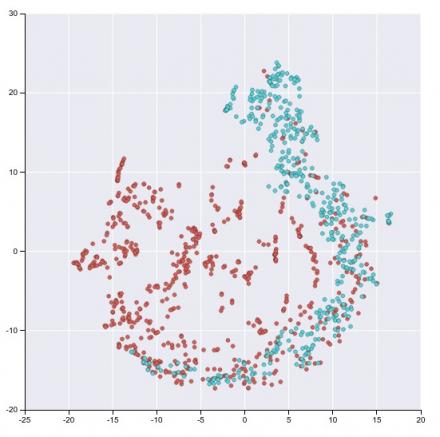
这是DDGK 学习的用于比较蛋白质的潜在表示的t-SNE可视化。蓝点表示编码酶的蛋白质,红点表示不编码酶的蛋白质。我们可以看到,编码与蛋白质的结构特性相关(无论它是否编码酶),即使在训练期间没有提供此上下文。(请注意,这是表示的投影,因此绝对轴值没有意义。)
在上面的例子中,我们展示了这些表示如何自动学习表示图并以编码其潜在功能相似性的方式对齐它们。在其他数据集上进行的实验表明,我们可以捕捉不同类型的图(语言、生物和社交互动)之间的相似性和差异性。
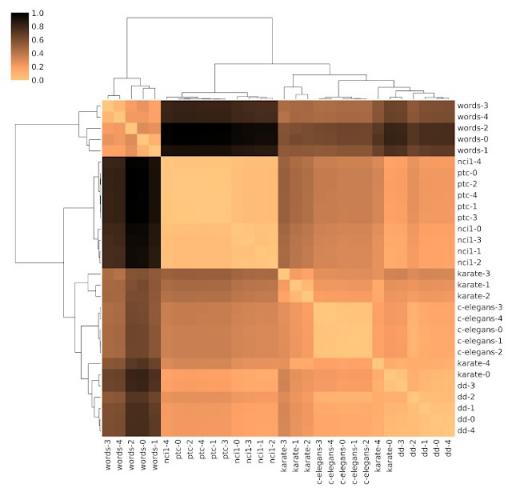
使用 DDGK 编码和对齐的不同数据集之间的成对距离。颜色表示潜在空间中的距离,相似度范围从 0(相同)到 1.0(非常不同)。我们看到,可以将表示聚类以将相似的数据集分组在一起 — 例如,数据集 nci1 和 ptc 都是化合物数据集。
快速准确地近似谱描述符
图的谱是一种强大的表示,它对其属性进行编码,包括图节点之间的连接模式和聚类信息。研究表明,谱可以传达关于不同对象属性的丰富信息,例如鼓声、 3D形状、图形和一般高维数据。谱图描述符的应用包括AutoML系统、动态图中的异常检测和化学分子表征。
目前,基于学习的系统(如 DDGK)无法扩展到大图或大图集合。或者,可以使用没有学习组件的谱信息来获得更理想的缩放属性。然而,计算大图的谱描述符在计算上是令人望而却步的。我们最近的论文通过提出 SLaQ(一种近似图描述符系列的方法)来解决这个问题。我们的方法使用随机近似算法来计算谱函数的轨迹,这使我们能够研究几个众所周知的谱图特征,如冯·诺依曼图熵、埃斯特拉达指数、图能量和NetLSD。
例如,我们使用 SLaQ 来监控维基百科图结构中的异常变化。SLaQ 使我们能够辨别页面图结构中的有意义的变化与诸如批量页面重命名之类的琐碎变化。我们的实验表明,近似精度平均提高了两个数量级。
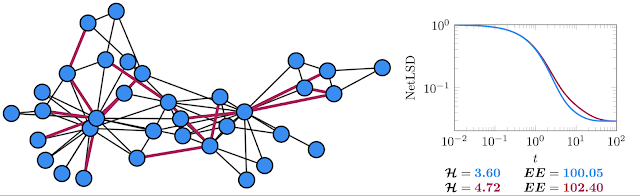
左图:著名的空手道图代表了两个武术俱乐部的社交互动。右图:计算原始图的谱描述符(NetLSD、VNGE 和埃斯特拉达指数)(蓝色)和删除边的版本(红色)。
结论
图的无监督表示学习是一个重要问题,我们相信我们在此重点介绍的方法是该领域令人兴奋的进步!具体来说,SLaQ使我们能够计算海量数据集的原则表示,而DDGK引入了一种自动学习数据集之间对齐的机制。我们希望我们的贡献将有助于推进大型数据集的分析,并有助于理解随时间变化的图数据集(如推荐系统中使用的数据集)的变化。
致谢我们感谢为这些工作做出贡献的 Marina Munkhoeva、Rami Al-Rfou 和 Dustin Zelle。有关图挖掘团队(算法和优化
组的一部分)的更多信息,请访问我们的页面。
评论