每个人的基因组由超过 30 亿个 DNA 字母组成,它们共同编码了每个人与生俱来的生化机制。然而,只有一小部分基因组(约 400-500 万个位置)在两个人之间有所不同。尽管如此,每个人独特的基因组都会与他们所经历的环境相互作用,从而决定他们的大部分健康结果。了解遗传变异与特征之间关系的一个关键方法是全基因组关联研究(GWAS),其中对队列中存在的每个遗传变异进行单独检查,以确定其与目标特征的相关性。GWAS 结果可用于通过识别与目标疾病密切相关的基因来识别和确定潜在治疗目标的优先顺序,也可用于建立多基因风险评分(PRS),以根据个体中存在的变异的综合影响来预测疾病倾向。然而,虽然准确测量个体的特征(称为表型分析)对于 GWAS 至关重要,但它通常需要精心的专家策划和/或主观判断。
在“基于大规模机器学习的表型分析显著改善视神经乳头形态的基因组发现”中,我们展示了如何使用机器学习 (ML) 模型对医学成像数据进行分类,从而改进 GWAS。我们描述了如何训练模型以针对表型生成特征预测,以及如何使用这些预测来识别新的遗传关联。然后,我们展示了发现的新关联提高了 PRS 准确性,并以青光眼为例,解剖眼部特征的改进与人类疾病有关。我们已在基因组学研究 GitHub 存储库中发布了模型训练代码及其使用的详细文档。
识别与眼部解剖特征相关的遗传变异
先前的研究表明,ML 模型可以识别眼部疾病、皮肤病和异常乳房 X 线照片结果,其准确度接近或超过领域专家的最新方法。由于识别疾病是表型分析的一个子集,我们推断 ML 模型可以广泛用于提高 GWAS 表型分析的速度和质量。
为了测试这一点,我们选择了一个使用眼底图像的模型来准确预测患者是否应该接受青光眼评估。该模型使用眼底图像来预测视神经乳头(视神经连接视网膜的区域)和视杯(视神经乳头中心的白色区域)的直径。这两个解剖特征的直径比(称为垂直杯盘比,或 VCDR)与青光眼风险密切相关。
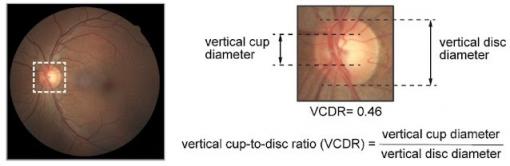
代表性的视网膜眼底图像显示垂直杯盘比,这是青光眼的重要诊断测量值。
我们应用该模型预测了英国生物库 中所有个人眼底图像的 VCDR ,英国生物库是全球最大的数据集,可供世界各地的研究人员进行公共健康相关研究,其中包含约 500,000 名假名(英国生物库的去身份化标准)个人的大量表型和基因数据。然后,我们在此数据集中执行了 GWAS,以确定与基于模型的 VCDR 预测相关的基因变异。
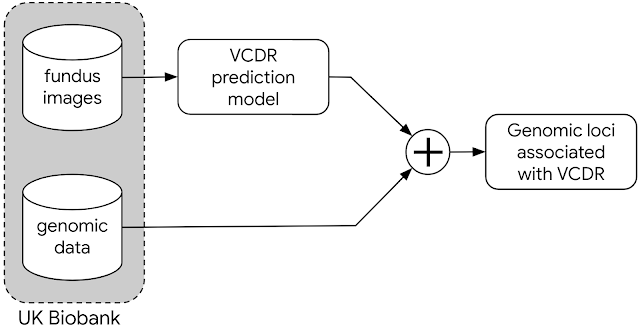
应用基于临床数据训练的 VCDR 预测模型来生成 VCDR 的预测值,从而发现 VCDR 特征的遗传关联。
基于 ML 的 GWAS 确定了 156 个与 VCDR 相关的不同基因组区域。我们将这些结果与另一个团队(Craig 等人,2020 年)对同一英国生物库数据进行的 VCDR GWAS 进行了比较,其中专家们精心标记了所有 VCDR 图像。基于 ML 的 GWAS 复制了 Craig等人发现的 65 个关联中的 62 个,这表明该模型准确预测了英国生物库图像中的 VCDR。此外,基于 ML 的 GWAS 发现了 93 个新的关联。
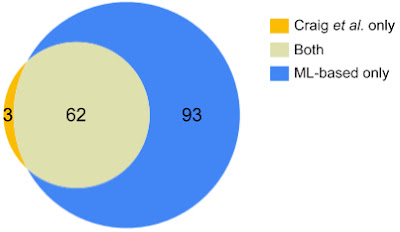
通过详尽的专家标记方法(Craig等人,左)和我们基于 ML 的方法(右)发现的具有统计学意义的 GWAS 关联的数量,中间是共享关联。
基于 ML 的 GWAS 改进了多基因模型预测
为了验证基于 ML 的 GWAS 中发现的新关联是否具有生物学相关性,我们使用 Craig等人和基于 ML 的 GWAS 结果开发了独立的 PRS,并在英国生物银行的一个子集以及一个完全独立的队列(EPIC-Norfolk)中测试了它们预测人类专家标记的 VCDR 的能力。在这两个数据集中,从基于 ML 的 GWAS 开发的 PRS 显示出比从专家标记方法构建的 PRS 更高的预测能力,这提供了强有力的证据表明基于 ML 的方法发现的新关联影响 VCDR 生物学,并表明模型表型准确性的提高(即更准确的 VCDR 测量)转化为更强大的 GWAS。
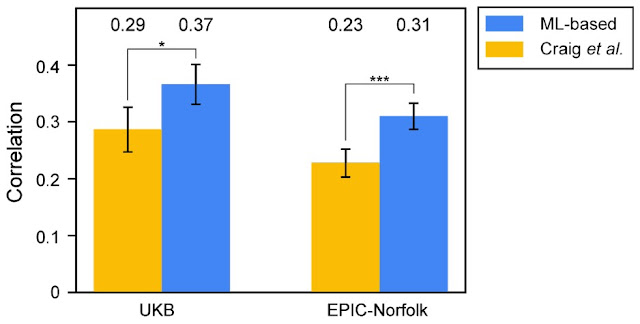
基于 ML 的方法与详尽专家标记方法 (Craig等人) 生成的 VCDR 多基因风险评分 (PRS) 之间的相关性。在这些图中,y 轴上的值越高,相关性就越大,因此仅从遗传数据进行的预测就越大。[* — p ≤ 0.05;*** — p ≤ 0.001]
作为第二项验证,因为我们知道 VCDR 与青光眼密切相关,我们还调查了基于 ML 的 PRS 是否与自报患有青光眼或有提示青光眼或青光眼治疗的医疗程序代码的个体相关。我们发现,使用我们的模型预测确定的 VCDR PRS 也可以预测个体患有青光眼的概率。在这个群体中,PRS 比平均值高出 2.5 个或更多标准差的个体患青光眼的可能性是其他人的 3 倍以上。我们还观察到,基于 ML 的表型的 VCDR PRS 比从广泛的手动表型分析中产生的 VCDR PRS 更能预测青光眼。
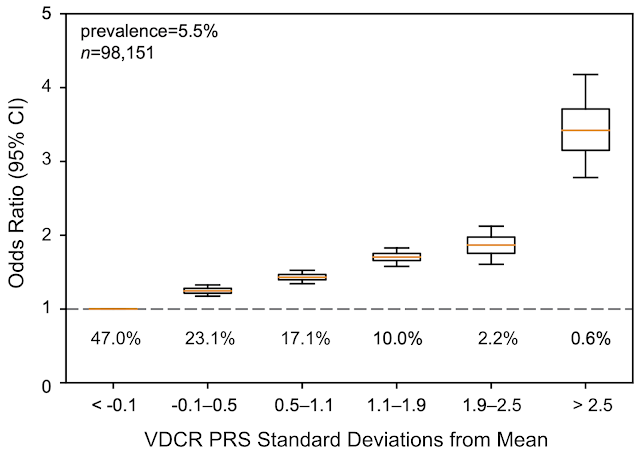
使用基于 ML 的表型确定的 VCDR 的 PRS 分层的青光眼(自我报告或 ICD 代码)几率比(以与平均值的标准差表示)。在此图中,y 轴显示个体患有青光眼的概率相对于基线率(用虚线表示)。x 轴显示与 PRS 平均值的标准差。数据以标准箱线图的形式显示,其中显示了平均值(橙色线)、第一和第三四分位数以及最小值和最大值。
结论
我们已经证明,机器学习模型可用于快速对 GWAS 的大群体进行表型分析,并且这些模型可以提高此类研究中的统计能力。虽然这些示例是根据视网膜成像预测的眼睛特征而展示的,但我们期待探索这一概念如何普遍应用于其他疾病和数据类型。
致谢
我们要特别感谢共同作者、Moorfields 眼科医院的Anthony Khawaja 博士贡献了他丰富的医学专业知识。我们还要感谢 Jamie Craig 教授及其同事的努力,他们对英国生物库图像进行了详尽的标记,这使我们能够与我们的方法进行比较。该研究的几位作者以及 Stuart MacGregor 教授及其在澳大利亚和 Max Kelsen 的合作者都独立复制了这些发现,我们也重视这些科学贡献。最后,这项工作总结了以下 Google 贡献者的工作,我们要感谢他们:Babak Alipanahi、Farhad Hormozdiari、Babak Behsaz、Justin Cosentino、Zachary R. McCaw、Emanuel Schorsch、D. Sculley、Elizabeth H. Dorfman、Sonia Phene、Naama Hammel、Andrew Carroll 和 Cory Y. McLean
评论